PathoSeq
Mattrygghet med høy presisjon – Patogen-genomikk for matindustrien

«PathoSeq» har som målsetning at bedrifter i norsk matindustri blir forberedt på å håndtere utfordringer og utnytte fordelene ved moderne DNA sekvenserings-teknologi for overvåkning og kontroll av matbårne patogene bakterier. Prosjektet fokuserer på Listeria monocytogenes, som er den viktigste mattrygghetsutfordringen i norsk matindustri.
Start
01. apr 2019
Slutt
31. mar 2023
Finansiert av
Norges forskningsråd
Samarbeid
I tillegg til Nofima deltar også forskningsmiljø fra Senter for Rettsinformatikk (SERI) ved Universitetet i Oslo og fra Unit of Food Microbiology ved University of Veterinary Medicine Vienna. Industripartnere som deltar i prosjektet er Nortura, Grilstad, Norsk Kylling, Orkla Foods, Kjøtt og Fjørfebransjens Landsforbund (KLF), SinkabergHansen, SalMar, Slakteriet, Sjømat Norge, Eurofins Genomics og Aquatiq Sense.
Prosjektleder(e):
Bakgrunn
Norsk næringsmiddelindustri driver et kontinuerlig kvalitetsarbeid for å møte utfordringer knyttet til mattrygghet, og moderne matproduksjon er avhengig av at man raskt finner og fjerner smittekilder for sykdomsfremkallende bakterier. Sentralt i dette arbeidet er å overvåke forekomst av uønskede bakterier i råvarer, produksjonsmiljø og produkter. Effektiv problemløsning og forebygging krever raskere og mer spesifikke påvisningsmetoder.
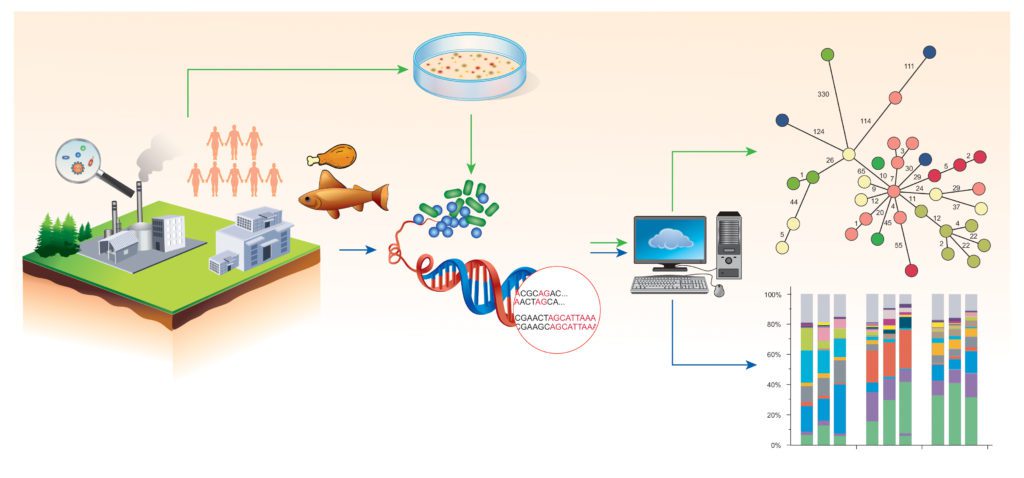
Helgenomsekvensering (whole genome sequencing; WGS) er en metode for å bestemme en organismes totale arvemateriale (DNA).
WGS-analyser er den mest sensitive og spesifikke metoden for å bestemme nivå av slektskap mellom for eksempel et pasientisolat og en bakterie funnet i mat eller matproduksjonsmiljø. Metodikken benyttes av folkehelse- og mattilsynsmyndigheter ved utbruddsetterforskning og rutinemessig overvåking av patogene bakterier i matkjeden. Dette kan gi mulighet til å spore bakterier som gir sykdom tilbake til enkeltanlegg.
Kunnskap og nytteverdi
I prosjektet jobber vi med å se på muligheter og barriærer knyttet bruk av WGS-metodikk i matindustrien. Hvis metoden brukes til mer effektiv kartlegging av smittekilder og smitteveier i bedriftenes forebyggende arbeid, vil man kunne sette inn tiltak før problemer oppstår eller vokser.
WGS-analyser gir også kunnskap om bakterienes funksjonelle egenskaper, som virulens, patogenisitet og antibiotikaresistens, noe som kan benyttes for å forbedre planer for risikobasert håndtering av mattrygghetspørsmål.
Resultater
Prosjektet har sekvensert nærmere 800 miljøisolater av Listeria monocytogenes fra 15 forskjellige prosesseringsanlegg for kjøtt og sjømat, samlet inn over en periode på over 30 år. Dette har gitt økt kunnskap om opphav, smitteveier, mangfold og distribusjon av ulike Listeria-varianter i norsk matindustri.
Kartlegging av slektskap mellom de individuelle isolatene viste at over halvparten var svært nært beslektet med bakterieisolater funnet i andre fabrikker. Likheten mellom disse var på nivå med det man ofte ser blant isolater knyttet til samme utbrudd av listeriose. Dette betyr at en bred oversikt over hvilke listeria-kloner som sirkulerer i norske matkjeder er viktig bakgrunnskunnskap ved tolking av WGS-resultater fra for eksempel etterforskning av matbårne utbrudd.
Undersøkelse av genetiske faktorer viste at de dominerende listeria-variantene som hadde etablert seg som husstammer i flere fabrikker var bedre tilpasset produksjonsmiljøet. Variantene som ble funnet flere steder hadde oftere gener som gir resistens mot forskjellige typer stress, inkludert renhold og desinfeksjon.
Videre har vi undersøkt om det er mulig å bruke WGS-data for å se etter spesielle gener som har betydning for alvorlighetsgrad ved sykdom, med tanke på å muligheten for å bruke denne informasjonen i risikovurderinger i matindustrien. Resultatene her viste at per i dag har denne fremgangsmåten begrensninger, siden de genetiske analysene ikke alltid korrelerte med eksperimentelle data for infeksjonseffektivitet bestemt ved undersøkelse av bakteriene i celletester.
Vi har sett nærmere på om metagenomiske sekvenseringsmetoder kan brukes til å gi raskere og mer sensitive prøvesvar, noe som kan gi bransjen mulighet til raskere å sette inn tiltak ved positive prøver. Resultatene viste at vi ved hjelp av Nanopore sekvenseringsteknologi kunne påvise listeria etter fire timer oppformering i selektivt medium. I tillegg kan metoden brukes til å påvise andre mikroorganismer fra prosessmiljøet. Sekvensering kan også brukes til å se om det er flere enn en variant av listeria tilstede i prøven.
Pratkiske analysestrategier og regulatoriske aspekter
Prosjektet har vært involvert i å utvikle dataverktøy til bruk i matindustrien for visualisering og håndtering av WGS-resultater. Vi vil videre utarbeide praktiske analysestrategier for hvordan sekvensdata kan brukes for å styrke bedriftenes internkontrollsystemer.
Prosjektet benytter en tverrfaglig tilnærming for å gi råd til matindustrien basert på vitenskapelige resultater. Dette vil inkludere å kartlegge juridiske og forvaltningsmessige aspekter som er relevante i forhold til implementering av WGS metodikk for regulering og kontroll av matsikkerhet, nasjonalt og internasjonalt. Dette arbeidet ledes av Universitetet i Oslo.